par Katerina Foka Sandoval et Valentin Gobert
NEW ! Improve your english
For the english version; have a look below.
Qu’est ce que la syphilis ?
La syphilis est une maladie vénérienne (sexuellement transmissible), qui peux être mortelle, causée par le Treponema pallidum ou tréponème.
La faculté du tréponème à modifier sa surface le rend difficile à combattre pour le système immunitaire qui peine à le reconnaître. Le détecter rapidement pour pouvoir traiter les patients devient alors primordial.

Le treponema pallidum
La fonction des anticorps :
Les anticorps produits par des cellules du système immunitaire (lymphocytes B) possèdent des parties variables qui leur permettent de reconnaître les corps étrangers, ou antigènes, comme les protéines de surface du tréponème, et s’y fixer (fig.1).
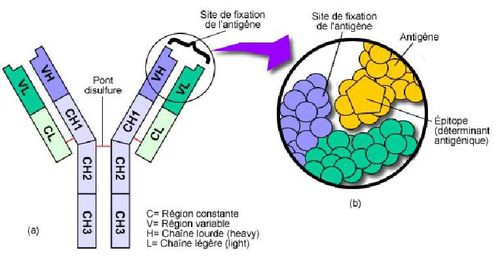 figure 1
figure 1
L’utilisation des anticorps dans le cas de l’immunofluorescence :
En laboratoire, les anticorps utilisés pour reconnaître les antigènes (par exemple les bactéries) sont appelés anticorps primaires.
On ajoute en plus des anticorps secondaires munis d’un composé fluorescent qui permet de le visualiser sous UV. Ces derniers se fixent sur l’anticorps primaire (voir fig 2 et fig.3) ce qui permet de mettre en lumière le composant étudié.
Cette technique peut être utilisée pour différentes bactéries si on possède les anticorps spécifiques capable de détecter leurs antigènes.
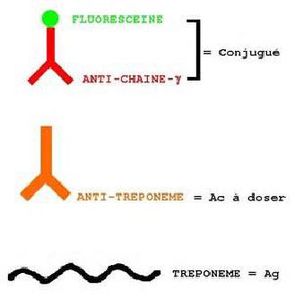
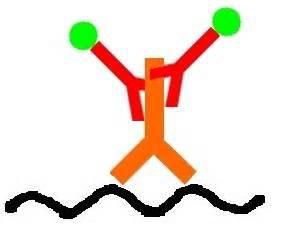
Figure 2 Figure 3
La visualisation du tréponème au microscope.
Le microscope à fluorescence est composé d’une lampe à arc qui émet une lumière qui, après avoir été filtrée, produit des UV. Les UV vont être projetés sur l’échantillon qui va être excité et les composés fluorescents de l’échantillon vont émettre de la lumière. Celui-ci sera donc plus visible au microscope.
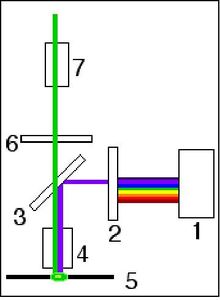
Le microscope à fluorescence : 1.Lampe à arc 2.Filtre d’excitation 3.Miroir dichroïque 4.Objectif 5.Préparation 6.Filtre d’émission 7.Oculaire
La visualisation du Tréponème peut se faire avec la technique présentée au-dessus, c’est à dire que nous prélevons le sérum d’un patient pour vérifier s'il contient ou non les anticorps spécifiques de la bactérie. Par la suite nous introduisons les anticorps secondaires qui vont venir se fixer sur les anticorps primaires. Ces anticorps secondaires portent une partie fluorescente.
Le mécanisme du microscope à fluorescence permet alors de visualiser les bactéries ainsi que d’estimer leur nombre et donc de conclure sur l’état de santé de la personne. Dans la photo, les tréponèmes sont bien visibles, et le patient à intérêt a se soigner !
![IFT Helico 03[1]](http://img.over-blog.com/500x364/5/22/79/75/IFT_Helico_03-1-.gif)
Et comme l’a si bien dit Sarah lors de notre visite à l’UNIGE, l’oeil collé à l’oculaire du microscope à fluorescence : « Whaouh, c’est trop stylé ! »
Crédit photos: BIOutils
Syphilis detection through the immunofluorescence technique
English version - Translation by Katerina Foka
What is syphilis ?
Syphilis is a venereal disease (sexually transmitted), that can result in death in some cases. It is induced by the bacteria Treponema pallidum also calledTreponem.
The ability it has of modifying its surface continually makes it very difficult to fight for the immune system which has some trouble recognizing it.
A fast detection method becomes essential, the patients needing to be treated as soon as possible.
The functions of antibodies:
The antibodies produced by the immune system’s cells (B lymphocytes) have two changing parts which allow them to recognize elements from foreign bodies, called antigens, like the surface proteins of the Treponem, and attach to it. (see fig.1).
The use of antibodies in immunofluorescence :
In a lab, the antibodies used to recognize different antigens (like the ones from bacteria) are called primary antibodies.
Once the primary ones have attached to the antigens the secondary antibodies who have a fluorescent component are added, this allows UV visualization later. The second antibodies are formatted to detect the first ones and attach to them (see fig.3) this is what helps shed light, quite literally, on the studied components.
This technique can be used to detect different bacteria if one has access to the specific antibodies capable of detecting their antigen.
Microscopic visualization of the treponem:
The fluorescence microscope is made up from a lamp whose filtered light produces UVs. These will then be projected on to the sample, The fluorescent components (which react to UV’s) will get excited and produce a visible light that can be observed through the lens. The bacteria are then visible.
Visualizing the Treponem can be achieved through this technique, the serum of the patient has first to be extracted. The secondary antibodies will then be introduced and attach to the primary ones created by the patients immune system. The secondary ones carry a fluorescent part.
The fluorescent’s microscope mechanism allows to visualize the bacteria and to estimate their number, and thus to determine the health state of the patient. In the picture above, the bacteria are well visible, and the patient should be treated fast !
/image%2F1527397%2F20210527%2Fob_860ead_pcr-image.png)








/image%2F1527397%2F20160610%2Fob_4e2c79_img-20151104-143954.jpg)


/image%2F1527397%2F20151117%2Fob_30bc64_img-20151104-143553.jpg)
/image%2F1527397%2F20151117%2Fob_266af7_img-20151104-145456.jpg)
/image%2F1527397%2F20151117%2Fob_0f8dab_img-20151104-145503.jpg)
/image%2F1527397%2F20151117%2Fob_e7a7f7_img-20151104-145833.jpg)
/image%2F1527397%2F20151117%2Fob_f64db2_img-20151104-152030.jpg)
/image%2F1527397%2F20151117%2Fob_e98f8f_img-20151104-161107.jpg)
/image%2F1527397%2F20151117%2Fob_480362_img-20151104-152321.jpg)
/image%2F1527397%2F20151117%2Fob_794fd6_img-20151104-143954.jpg)
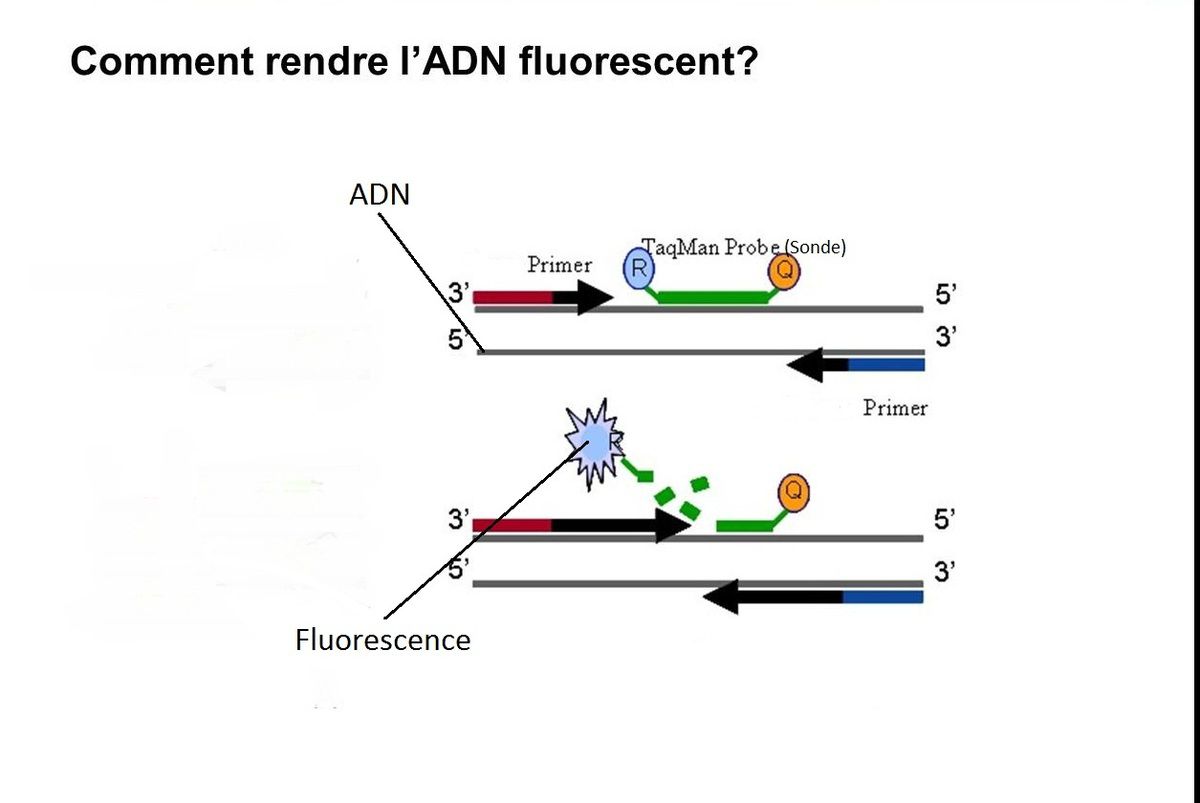


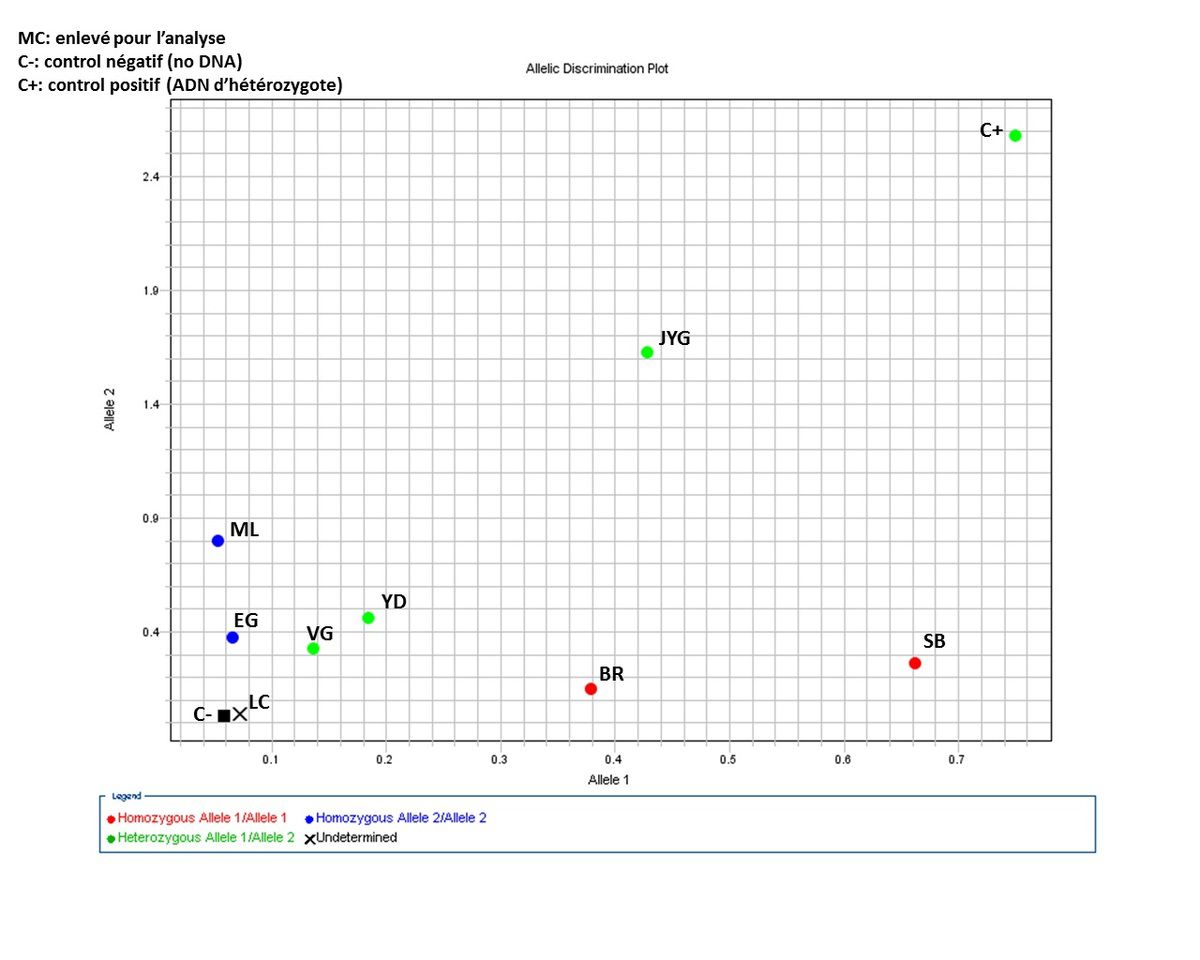






 figure 1
figure 1


![IFT Helico 03[1]](http://img.over-blog.com/500x364/5/22/79/75/IFT_Helico_03-1-.gif)